中国全科医学 ›› 2025, Vol. 28 ›› Issue (36): 4592-4604.DOI: 10.12114/j.issn.1007-9572.2024.0418
所属专题: 肿瘤最新文章合辑
收稿日期:2024-08-13
修回日期:2025-09-13
出版日期:2025-12-20
发布日期:2025-12-04
通讯作者:
熊丹
作者贡献:
谭洁文负责研究的构思与设计,研究的实施,撰写论文;陈嫦、钟锦漫进行数据的收集与整理,统计学处理,图表的绘制;胡婉贞、白海进行论文的修订;熊丹负责文章审查,对文章整体负责,监督管理。
基金资助:
TAN Jiewen, CHEN Chang, ZHONG Jinman, HU Wanzhen, BAI Hai, XIONG Dan*( )
)
Received:2024-08-13
Revised:2025-09-13
Published:2025-12-20
Online:2025-12-04
Contact:
XIONG Dan
摘要: 背景 乳酸和乳酸代谢在一些实体肿瘤发生和进展中的意义已有报道。然而,乳酸代谢在多发性骨髓瘤(MM)患者预后及肿瘤微环境(TME)中的作用尚不清楚。 目的 本研究旨在构建基于乳酸代谢的MM患者风险评分预后模型,并探索乳酸代谢对TME的影响。 方法 首先从基因表达综合数据库(GEO)下载MM数据集GSE136324、GSE4581的转录组测序表达数据和样本生存信息。从MSigDB数据库和GeneCards数据库筛选出纳入后续分析的乳酸代谢相关基因。应用一致性聚类和Cibersort方法探索乳酸代谢相关基因与TME的关系。采用Wilcoxon秩和检验分析MM样本的TME差异。基于线性回归模型筛选出与生存预后关联的乳酸代谢相关基因,并利用森林图和Kaplan-Meier生存曲线进行可视化呈现,生存曲线的比较采用Log-rank检验。采用Lasso回归分析筛选变量,采用单因素和多因素Cox回归分析筛选与预后相关的乳酸代谢相关基因建立预后模型。采用Kaplan-Meier生存分析以及受试者工作特征(ROC)曲线评估模型的预测能力。采用Pearson相关性分析探讨乳酸代谢模型风险评分与免疫评分、基质评分和肿瘤纯度的相关性。 结果 基于乳酸代谢相关基因的表达谱应用ConsensusClusterPlus包进行一致性聚类,识别出2个乳酸代谢相关基因亚类Lactate.clusterA和Lactate.clusterB,Lactate.clusterA亚类预后较Lactate.clusterB亚类预后更差(χ2=19.11,P<0.000 1)。初始B细胞、记忆B细胞、浆细胞、CD8阳性T细胞、静息记忆CD4阳性T细胞、活化记忆CD4阳性T细胞、滤泡辅助性T细胞、γδT细胞、静息自然杀伤细胞、活化自然杀伤细胞、单核细胞、M0型巨噬细胞、M2型巨噬细胞、活化树突状细胞、静息肥大细胞、活化肥大细胞、嗜酸性粒细胞、中性粒细胞在两类亚类样本间差异有统计学意义(P<0.05)。筛选出预后相关的乳酸代谢相关基因,胆碱磷酸转移酶(PC)、聚合酶γ(POLG)、脂肪酸合酶(FASN)、脂肪酸结合蛋白2(FABP2)、溶质载体家族7成员5(SLC7A5)、分支链氨基酸转氨酶2(BCAT2)、单羧酸转运蛋白8(SLC16A8)、3-羟基酰基辅酶A脱氢酶(HAGH)、6-磷酸果糖激酶(PFKL)、3-磷酸甘油酸激酶2(PGK2)、丙酮酸激酶同工酶R(PKLR)、碳酸酐酶2(CA2)、葡萄糖转运蛋白1(SLC2A1)基因高表达预后较好;丙酮酸脱氢酶磷酸酶催化亚基1(PDP1)、乙酰辅酶A琥珀酰转移酶(AGK)、过氧化物酶体组装蛋白12(PEX12)和6-磷酸果糖-2-激酶/果糖-2,6-双磷酸酶2(PFKFB2)基因高表达预后较差(P<0.05)。筛选出SLC2A1、CA2、PKLR、PFKL、PFKFB2、SLC16A8、SLC7A5、AGK、FABP2、POLG、PC 11个独立预测因子变量纳入多因素Cox回归分析构建预测模型,根据风险得分的中位数将样本分为高风险组与低风险组,训练集高风险组生存率低于低风险组(χ2=59.02,P<0.05)。绘制ROC曲线,模型预测5年生存的ROC曲线下面积(AUC)为0.781(95%CI=0.664~0.886)。验证集高风险组生存率低于低风险组(χ2=9.24,P<0.05),模型预测1年生存的AUC为0.64(95%CI=0.542~0.737)。收集训练数据集样本的免疫治疗效果资料,结果显示高风险组与低风险组预后情况存在差异(Z=-2.469,P=0.014),高风险组是患者预后的影响因素(P<0.05)。比较高风险组、低风险组样本免疫细胞浸润水平,浆细胞、CD8阳性T细胞、静息记忆CD4阳性T细胞、活化记忆CD4阳性T细胞、滤泡辅助性T细胞、静息自然杀伤细胞、活化自然杀伤细胞、单核细胞、M0型巨噬细胞、静息树突状细胞、活化树突状细胞、静息肥大细胞、活化肥大细胞、嗜酸性粒细胞丰度差异有统计学意义(P<0.05)。高风险组、低风险组样本甲型肝炎病毒细胞受体2(HAVCR2)、程序性细胞死亡1配体1(CD274)、脊髓灰质炎病毒受体(PVR)、CD80、细胞毒性T淋巴细胞相关抗原4(CTLA4)、程序性细胞死亡蛋白1(PDCD1)、CD200受体1(CD200R1)、CD276、CD200、B和T淋巴细胞衰减因子(BTLA)、半乳糖凝集素3(LGALS3)、V域免疫球蛋白抑制剂1(VTCN1)基因表达差异有统计学意义(P<0.05)。计算50 hallmark通路的富集得分,采用Pearson相关性分析探究乳酸代谢预后模型风险得分和不同通路的相关性,发现WNT beta catenin signaling、Androgen response和UV response通路与乳酸代谢预后模型风险得分呈正相关,KRAS signaling、Pancreas beta cells和Heme metabolism与乳酸代谢预后模型风险得分呈负相关(P<0.05);乳酸代谢预后模型风险得分与基质评分呈正相关(P<0.05),与肿瘤纯度无相关性(P>0.05)。 结论 本研究构建了一个基于乳酸代谢的MM患者风险评分预后模型,在预测长期生存方面表现更好。而乳酸代谢与TME相关分析提示乳酸代谢可能影响MM患者TME中的免疫细胞群,从而影响肿瘤的进展,并进而影响MM患者的预后。
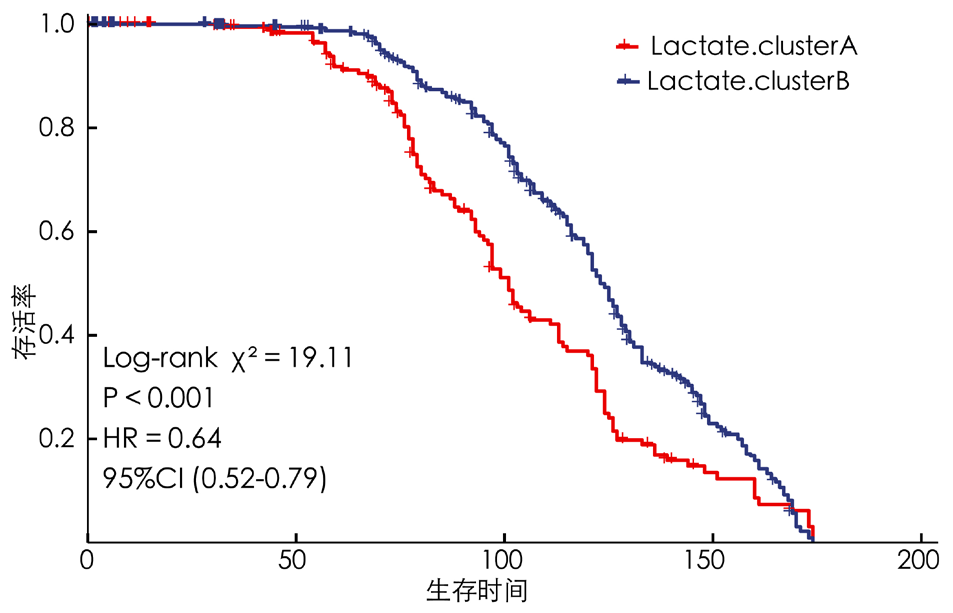
图2 Lactate.clusterA亚类与Lactate.clusterB亚类患者预后的Kaplan-Meier生存曲线图
Figure 2 Kaplan-Meier survival curves for prognosis of patients in the Lactate.cluster A subclass and the Lactate.Cluster B subclass

图3 Lactate.clusterA亚类与Lactate.clusterB亚类患者TME免疫细胞浸润差异热图
Figure 3 Heat map of the difference in TME immune cell infiltration between Lactate.cluster A subclass and Lactate.cluster B subclass patients
| 类别 | 例数 | 初始B细胞 | 记忆B细胞 | 浆细胞 | CD8阳性T细胞 | 初始CD4阳性T细胞 | 静息记忆CD4阳性T细胞 | 活化记忆CD4阳性T细胞 |
|---|---|---|---|---|---|---|---|---|
| Lactate.clusterA亚类 | 217 | 0.000(0.000,0.000) | 0.059(0.029,0.097) | 0.261(0.150,0.409) | 0.049(0.025,0.075) | 0.000(0.000,0.031) | 0.000(0.000,0.003) | 0.002(0.000,0.013) |
| Lactate.clusterB亚类 | 649 | 0.000(0.000,0.016) | 0.014(0.000,0.038) | 0.101(0.067,0.160) | 0.060(0.039,0.085) | 0.005(0.000,0.024) | 0.000(0.000,0.000) | 0.009(0.000,0.017) |
| Z值 | 48 512.5 | 110 646.0 | 114 533.0 | 56 905.0 | 69 105.0 | 75 791.0 | 57 997.0 | |
| P值 | <0.001 | <0.001 | <0.001 | <0.001 | 0.667 | 0.020 | <0.001 | |
| 类别 | 滤泡辅助性T细胞 | 调节性T细胞 | γδT细胞 | 静息自然杀伤细胞 | 活化自然杀伤细胞 | 单核细胞 | M0型巨噬细胞 | |
| Lactate.clusterA亚类 | 0.000(0.000,0.008) | 0.013(0.005,0.022) | 0.046(0.019,0.083) | 0.012(0.000,0.036) | 0.000(0.000,0.000) | 0.147(0.053,0.256) | 0.054(0.031,0.073) | |
| Lactate.clusterB亚类 | 0.000(0.000,0.003) | 0.012(0.002,0.024) | 0.062(0.034,0.091) | 0.001(0.000,0.022) | 0.000(0.000,0.005) | 0.330(0.260,0.383) | 0.065(0.046,0.089) | |
| Z值 | 82 396.0 | 73 363.5 | 59 753.0 | 83 366.0 | 62 058.0 | 24 191.0 | 53 436.0 | |
| P值 | <0.001 | 0.354 | <0.001 | <0.001 | 0.001 | <0.001 | <0.001 | |
| 类别 | M1型巨噬细胞 | M2型巨噬细胞 | 静息树突状细胞 | 活化树突状细胞 | 静息肥大细胞 | 活化肥大细胞 | 嗜酸性粒细胞 | 中性粒细胞 |
| Lactate.clusterA亚类 | 0.005(0.001,0.018) | 0.075(0.047,0.118) | 0.000(0.000,0.000) | 0.007(0.001,0.013) | 0.016(0.000,0.041) | 0.015(0.002,0.041) | 0.005(0.000,0.019) | 0.037(0.016,0.067) |
| Lactate.clusterB亚类 | 0.006(0.002,0.014) | 0.065(0.044,0.098) | 0.000(0.000,0.000) | 0.000(0.000,0.005) | 0.057(0.032,0.083) | 0.000(0.000,0.010) | 0.008(0.000,0.023) | 0.083(0.055,0.111) |
| Z值 | 69 405.0 | 79 771.0 | 71 939.0 | 98 554.0 | 30 518.0 | 101 090.5 | 63 498.0 | 33 555.0 |
| P值 | 0.751 | 0.003 | 0.080 | <0.001 | <0.001 | <0.001 | 0.027 | <0.001 |
表1 Lactate.clusterA亚类与Lactate.clusterB亚类患者TME免疫细胞浸润的CIBERSORT评分比较[M(P25,P75)]
Table 1 Comparison of CIBERSORT scores for TME immune cell infiltration between Lactate.Cluster A subclass and Lactate.Cluster B subclass patients
| 类别 | 例数 | 初始B细胞 | 记忆B细胞 | 浆细胞 | CD8阳性T细胞 | 初始CD4阳性T细胞 | 静息记忆CD4阳性T细胞 | 活化记忆CD4阳性T细胞 |
|---|---|---|---|---|---|---|---|---|
| Lactate.clusterA亚类 | 217 | 0.000(0.000,0.000) | 0.059(0.029,0.097) | 0.261(0.150,0.409) | 0.049(0.025,0.075) | 0.000(0.000,0.031) | 0.000(0.000,0.003) | 0.002(0.000,0.013) |
| Lactate.clusterB亚类 | 649 | 0.000(0.000,0.016) | 0.014(0.000,0.038) | 0.101(0.067,0.160) | 0.060(0.039,0.085) | 0.005(0.000,0.024) | 0.000(0.000,0.000) | 0.009(0.000,0.017) |
| Z值 | 48 512.5 | 110 646.0 | 114 533.0 | 56 905.0 | 69 105.0 | 75 791.0 | 57 997.0 | |
| P值 | <0.001 | <0.001 | <0.001 | <0.001 | 0.667 | 0.020 | <0.001 | |
| 类别 | 滤泡辅助性T细胞 | 调节性T细胞 | γδT细胞 | 静息自然杀伤细胞 | 活化自然杀伤细胞 | 单核细胞 | M0型巨噬细胞 | |
| Lactate.clusterA亚类 | 0.000(0.000,0.008) | 0.013(0.005,0.022) | 0.046(0.019,0.083) | 0.012(0.000,0.036) | 0.000(0.000,0.000) | 0.147(0.053,0.256) | 0.054(0.031,0.073) | |
| Lactate.clusterB亚类 | 0.000(0.000,0.003) | 0.012(0.002,0.024) | 0.062(0.034,0.091) | 0.001(0.000,0.022) | 0.000(0.000,0.005) | 0.330(0.260,0.383) | 0.065(0.046,0.089) | |
| Z值 | 82 396.0 | 73 363.5 | 59 753.0 | 83 366.0 | 62 058.0 | 24 191.0 | 53 436.0 | |
| P值 | <0.001 | 0.354 | <0.001 | <0.001 | 0.001 | <0.001 | <0.001 | |
| 类别 | M1型巨噬细胞 | M2型巨噬细胞 | 静息树突状细胞 | 活化树突状细胞 | 静息肥大细胞 | 活化肥大细胞 | 嗜酸性粒细胞 | 中性粒细胞 |
| Lactate.clusterA亚类 | 0.005(0.001,0.018) | 0.075(0.047,0.118) | 0.000(0.000,0.000) | 0.007(0.001,0.013) | 0.016(0.000,0.041) | 0.015(0.002,0.041) | 0.005(0.000,0.019) | 0.037(0.016,0.067) |
| Lactate.clusterB亚类 | 0.006(0.002,0.014) | 0.065(0.044,0.098) | 0.000(0.000,0.000) | 0.000(0.000,0.005) | 0.057(0.032,0.083) | 0.000(0.000,0.010) | 0.008(0.000,0.023) | 0.083(0.055,0.111) |
| Z值 | 69 405.0 | 79 771.0 | 71 939.0 | 98 554.0 | 30 518.0 | 101 090.5 | 63 498.0 | 33 555.0 |
| P值 | 0.751 | 0.003 | 0.080 | <0.001 | <0.001 | <0.001 | 0.027 | <0.001 |
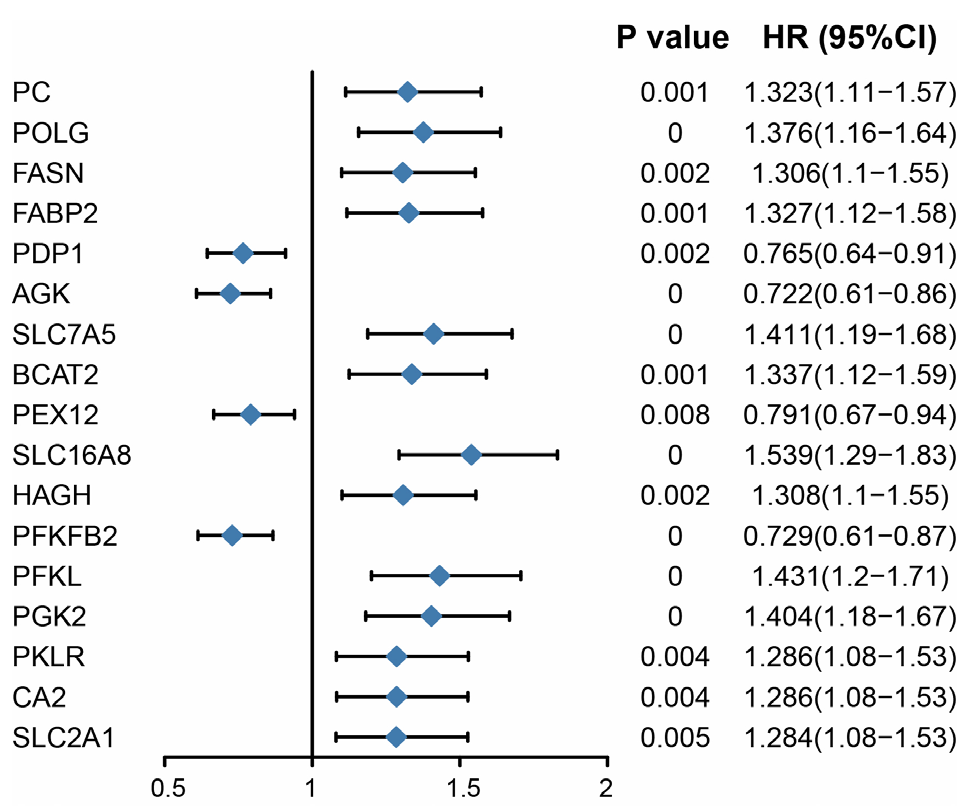
图4 预后显著相关乳酸代谢基因的森林图注:PC=胆碱磷酸转移酶,POLG=聚合酶γ,FASN=脂肪酸合酶,FABP2=脂肪酸结合蛋白2,SLC7A5=溶质载体家族7成员5,BCAT2=分支链氨基酸转氨酶2,SLC16A8=单羧酸转运蛋白8,HAGH=3-羟基酰基辅酶A脱氢酶,PFKL=6-磷酸果糖激酶,PGK2=3-磷酸甘油酸激酶2,PKLR=丙酮酸激酶同工酶R,CA2=碳酸酐酶2,SLC2A1=葡萄糖转运蛋白1,PDP1=丙酮酸脱氢酶磷酸酶催化亚基1,AGK=乙酰辅酶A琥珀酰转移酶,PEX12=过氧化物酶体组装蛋白12,PFKFB2=6-磷酸果糖-2-激酶/果糖-2,6-双磷酸酶2。
Figure 4 Forest map of lactic acid metabolism genes significantly related to prognosis
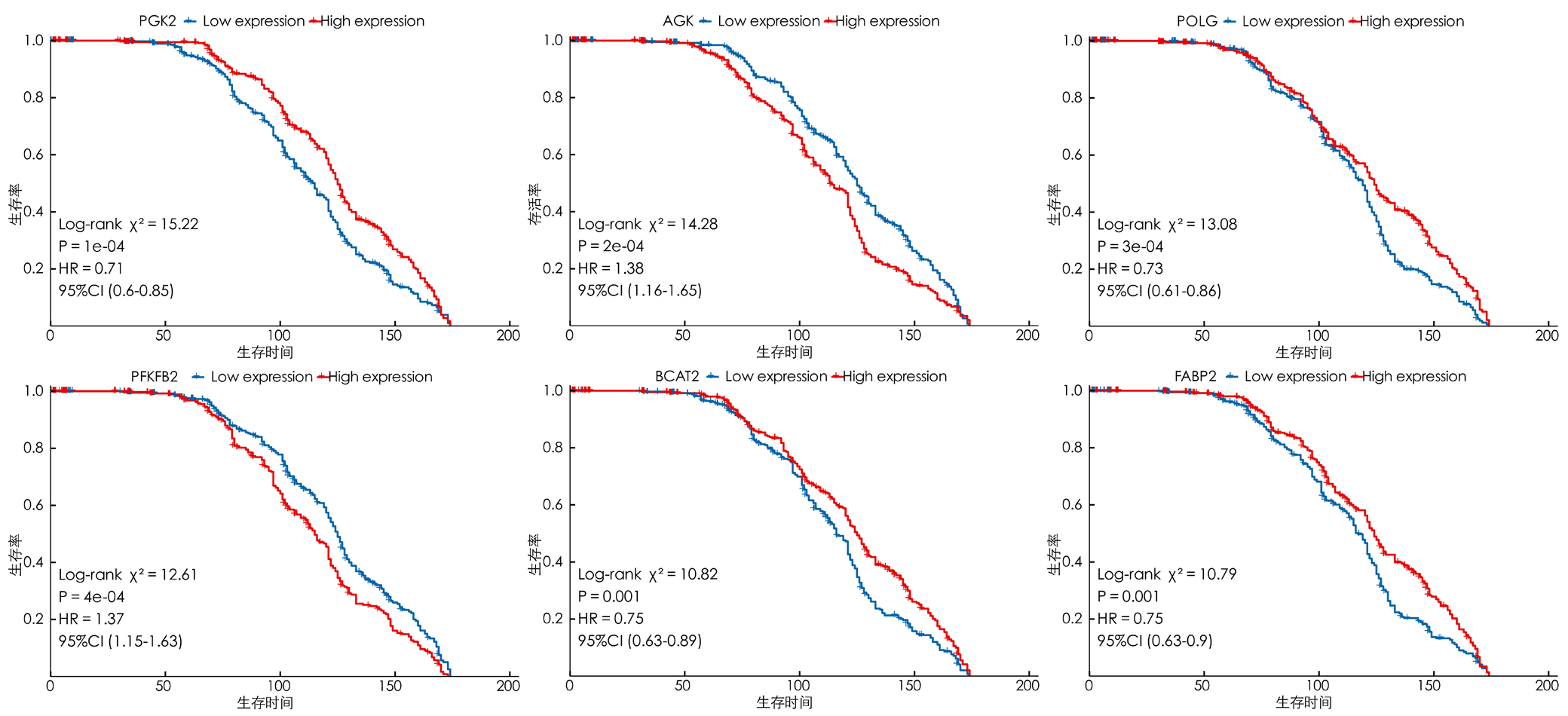
图5 生存显著相关前6位的乳酸代谢相关基因的Kaplan-Meier生存曲线图注:A~F分别为PGK2、AGK、POLG、PFKFB2、BCAT2、FABP2高、低表达组患者的生存曲线,蓝色曲线为低表达组,红色曲线为高表达组。
Figure 5 Kaplan-Meier survival curves of the top 6 lactic acid metabolism-related genes significantly associated with survival
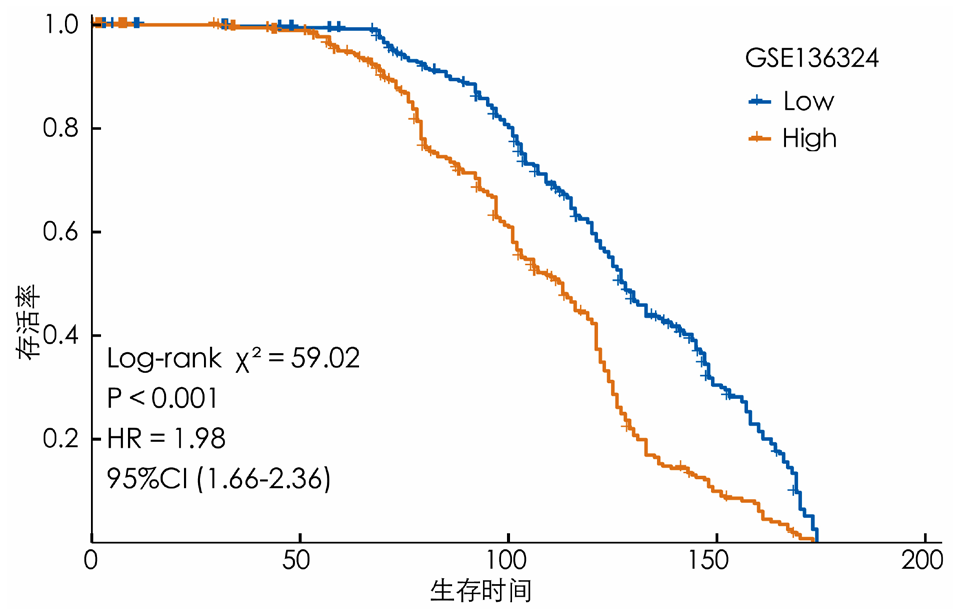
图9 训练数据集高风险组与低风险组患者预后的Kaplan-Meier生存曲线图
Figure 9 Kaplan-Meier survival curves of prognosis for patients in the high-risk group and the low-risk group of the training dataset
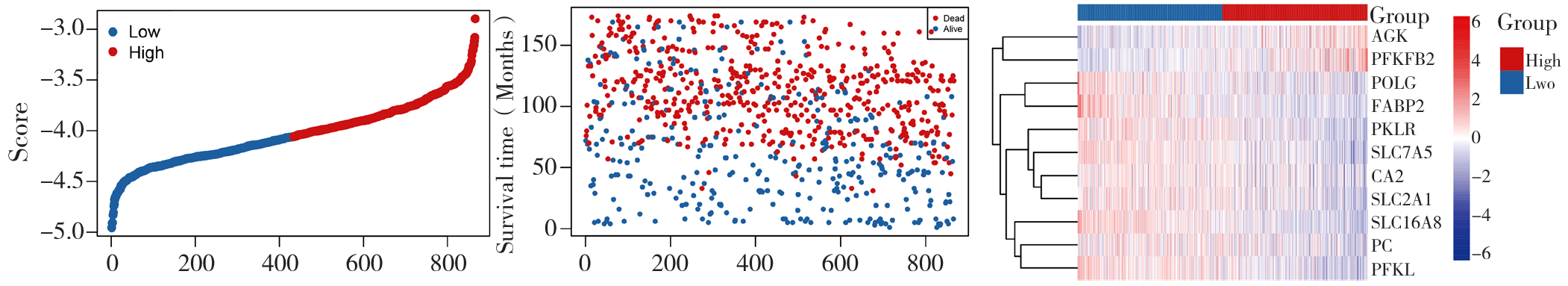
图11 预测模型基因分布与患者生存情况的三联图注:A为基于模型的样本打分图,B为用于展示生存时间(月)与样本的关系,C为热图展示了不同样本在多个基因上的表达水平。
Figure 11 Triple plot of gene distribution of the prediction model and patient survival
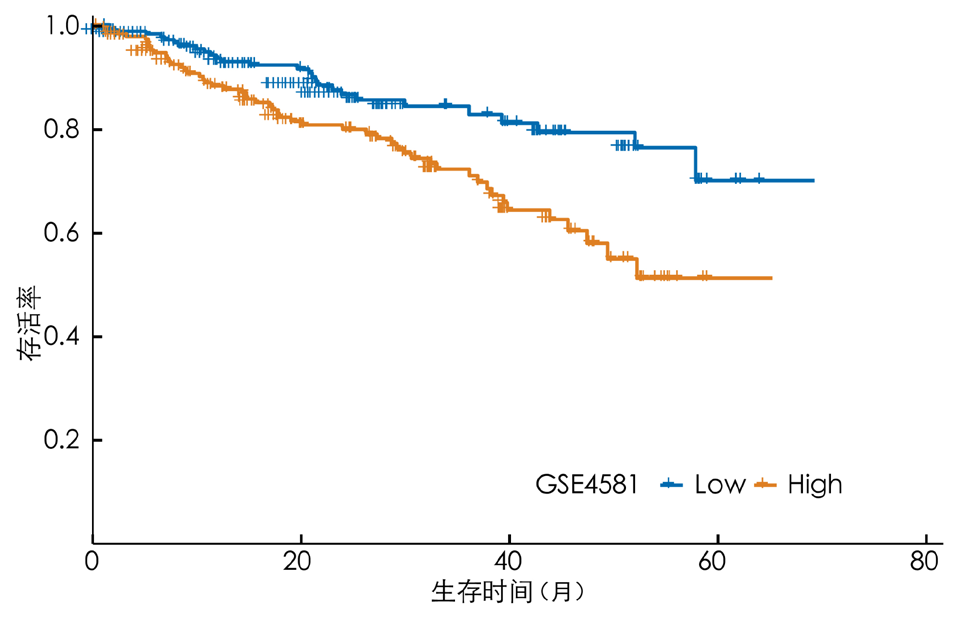
图12 验证数据集高风险组与低风险组患者预后的Kaplan-Meier生存曲线图
Figure 12 Kaplan-Meier survival curves for the prognosis of patients in the high-risk and low-risk groups in the validation dataset
| 组别 | 例数 | CR | PR | SD | PD |
|---|---|---|---|---|---|
| 高风险组 | 427 | 223(52.22) | 50(11.71) | 44(10.30) | 110(25.76) |
| 低风险组 | 432 | 23(59.26) | 23(5.32) | 53(12.27) | 100(23.15) |
表2 训练数据集高风险组与低风险组患者免疫治疗预后情况[例(%)]
Table 2 Prognosis of immunotherapy in high-risk and low-risk groups of patients in the training dataset
| 组别 | 例数 | CR | PR | SD | PD |
|---|---|---|---|---|---|
| 高风险组 | 427 | 223(52.22) | 50(11.71) | 44(10.30) | 110(25.76) |
| 低风险组 | 432 | 23(59.26) | 23(5.32) | 53(12.27) | 100(23.15) |
| 分组 | 例数 | 预后模型风险得分 |
|---|---|---|
| CR组 | 479 | -4.056 7 |
| PR组 | 73 | -3.918 2a |
| SD组 | 97 | -4.030 7 |
| PD组 | 210 | -4.032 6 |
| F值 | 4.635 | |
| P值 | 0.003 |
表3 训练数据集不同免疫治疗预后分组患者预后模型风险得分比较(分)
Table 3 Comparison of risk scores of prognosis models for patients in different immunotherapy prognosis groups in the training dataset
| 分组 | 例数 | 预后模型风险得分 |
|---|---|---|
| CR组 | 479 | -4.056 7 |
| PR组 | 73 | -3.918 2a |
| SD组 | 97 | -4.030 7 |
| PD组 | 210 | -4.032 6 |
| F值 | 4.635 | |
| P值 | 0.003 |
| 性别 | 例数 | 预后模型风险得分 |
|---|---|---|
| 男 | 533 | -4.046 2 |
| 女 | 326 | -4.019 6 |
| t值 | 1.243 | |
| P值 | 0.214 |
表4 训练数据集不同性别患者预后模型风险得分比较(分)
Table 4 Comparison of risk scores of prognostic models for patients of different genders in the training dataset
| 性别 | 例数 | 预后模型风险得分 |
|---|---|---|
| 男 | 533 | -4.046 2 |
| 女 | 326 | -4.019 6 |
| t值 | 1.243 | |
| P值 | 0.214 |
| 分组 | 例数 | 预后模型风险得分(分) |
|---|---|---|
| ≤65岁组 | 632 | -4.052 8 |
| >65岁组 | 227 | -3.989 4 |
| t值 | -2.655 | |
| P值 | 0.008 |
表5 训练数据集不同年龄分组患者预后模型风险得分比较(分)
Table 5 Comparison of prognostic model risk scores for patients in different age groups of the training dataset
| 分组 | 例数 | 预后模型风险得分(分) |
|---|---|---|
| ≤65岁组 | 632 | -4.052 8 |
| >65岁组 | 227 | -3.989 4 |
| t值 | -2.655 | |
| P值 | 0.008 |
| 变量 | 单因素分析 | 多因素分析 | ||
|---|---|---|---|---|
| HR(95%CI) | P值 | HR(95%CI) | P值 | |
| 高风险组 | 3.139(2.060~4.784) | <0.001 | 3.131(2.046~4.789) | <0.001 |
| >65岁 | 1.535(0.951~2.476) | 0.079 | 1.418(0.875~2.297) | 0.157 |
| 男性 | 0.782(0.518~1.182) | 0.244 | 0.781(0.515~1.185) | 0.246 |
表6 训练数据集治疗无效组患者预后影响因素的单因素与多因素Cox回归分析
Table 6 Univariate and multivariate Cox regression analyses of prognostic factors in the treatment ineffective group of patients in the training dataset
| 变量 | 单因素分析 | 多因素分析 | ||
|---|---|---|---|---|
| HR(95%CI) | P值 | HR(95%CI) | P值 | |
| 高风险组 | 3.139(2.060~4.784) | <0.001 | 3.131(2.046~4.789) | <0.001 |
| >65岁 | 1.535(0.951~2.476) | 0.079 | 1.418(0.875~2.297) | 0.157 |
| 男性 | 0.782(0.518~1.182) | 0.244 | 0.781(0.515~1.185) | 0.246 |
| 变量 | 单因素分析 | 多因素分析 | ||
|---|---|---|---|---|
| HR(95%CI) | P值 | HR(95%CI) | P值 | |
| 高风险组 | 1.844(1.514~2.246) | <0.001 | 1.849(1.517~2.253) | <0.001 |
| >65岁 | 0.978(0.763~1.253) | 0.858 | 0.94(0.731~1.209) | 0.629 |
| 男性 | 0.974(0.797~1.190) | 0.797 | 0.989(0.808~1.212) | 0.918 |
表7 训练数据集患者治疗有效组预后影响因素的单因素与多因素Cox回归分析
Table 7 Univariate and multivariate Cox regression analyses of prognostic factors in the effective treatment group of patients in the training dataset
| 变量 | 单因素分析 | 多因素分析 | ||
|---|---|---|---|---|
| HR(95%CI) | P值 | HR(95%CI) | P值 | |
| 高风险组 | 1.844(1.514~2.246) | <0.001 | 1.849(1.517~2.253) | <0.001 |
| >65岁 | 0.978(0.763~1.253) | 0.858 | 0.94(0.731~1.209) | 0.629 |
| 男性 | 0.974(0.797~1.190) | 0.797 | 0.989(0.808~1.212) | 0.918 |
| 组别 | 例数 | 初始B细胞 | 记忆B细胞 | 浆细胞 | CD8阳性T细胞 | 初始CD4阳性T细胞 | 静息记忆CD4阳性T细胞 | 活化记忆CD4阳性T细胞 |
|---|---|---|---|---|---|---|---|---|
| 低风险组 | 432 | 0.000(0.000,0.166) | 0.020(0.000,0.323) | 0.111(0.004,0.604) | 0.063(0.000,0.306) | 0.005(0.000,0.155) | 0.000(0.000,0.068) | 0.004(0.000,0.064) |
| 高风险组 | 427 | 0.000(0.000,0.228) | 0.024(0.000,0.480) | 0.139(0.003,0.793) | 0.053(0.000,0.239) | 0.004(0.000,0.116) | 0.000(0.000,0.083) | 0.010(0.000,0.053) |
| 检验统计量值 | 0.402 | -1.447 | -3.380 | 4.338 | -0.513 | -4.807 | -3.679 | |
| P值 | 0.69 | 0.15 | <0.01 | <0.01 | 0.61 | <0.01 | <0.01 | |
| 组别 | 滤泡辅助性T细胞 | 调节性T细胞 | γδT细胞 | 静息自然杀伤细胞 | 活化自然杀伤细胞 | 单核细胞 | M0型巨噬细胞 | |
| 低风险组 | 0.000(0.000,0.054) | 0.012(0.000,0.098) | 0.056(0.000,0.260) | 0.000(0.000,0.154) | 0.000(0.000,0.141) | 0.314(0.000,0.559) | 0.066(0.000,0.307) | |
| 高风险组 | 0.000(0.000,0.062) | 0.012(0.000,0.101) | 0.061(0.000,0.268) | 0.012(0.000,0.093) | 0.000(0.000,0.057) | 0.273(0.000,0.545) | 0.057(0.000,0.385) | |
| 检验统计量值 | 2.298 | 1.772 | -1.770 | -5.801 | 8.936 | 4.857 | 3.793 | |
| P值 | 0.02 | 0.08 | 0.08 | <0.01 | <0.01 | <0.01 | <0.01 | |
| 组别 | M1型巨噬细胞 | M2型巨噬细胞 | 静息树突状细胞 | 活化树突状细胞 | 静息肥大细胞 | 活化肥大细胞 | 嗜酸性粒细胞 | 中性粒细胞 |
| 低风险组 | 0.006(0.000,0.058) | 0.065(0.000,0.329) | 0.000(0.000,0.019) | 0.001(0.000,0.030) | 0.054(0.000,0.208) | 0.000(0.000,0.155) | 0.006(0.000,0.138) | 0.073(0.000,0.223) |
| 高风险组 | 0.006(0.000,0.149) | 0.069(0.000,0.373) | 0.000(0.000,0.072) | 0.003(0.000,0.037) | 0.042(0.000,0.244) | 0.002(0.000,0.143) | 0.008(0.000,0.142) | 0.073(0.000,0.318) |
| 检验统计量值 | 0.105 | -0.936 | -2.150 | -3.350 | 4.766 | -2.012 | -2.677 | 0.395 |
| P值 | 0.92 | 0.35 | 0.03 | <0.01 | <0.01 | 0.04 | <0.01 | 0.69 |
表8 风险高、低组样本免疫细胞浸润水平的差异[M(P25,P75)]
Table 8 Differences in immune cell infiltration levels between high-risk and low-risk group samples
| 组别 | 例数 | 初始B细胞 | 记忆B细胞 | 浆细胞 | CD8阳性T细胞 | 初始CD4阳性T细胞 | 静息记忆CD4阳性T细胞 | 活化记忆CD4阳性T细胞 |
|---|---|---|---|---|---|---|---|---|
| 低风险组 | 432 | 0.000(0.000,0.166) | 0.020(0.000,0.323) | 0.111(0.004,0.604) | 0.063(0.000,0.306) | 0.005(0.000,0.155) | 0.000(0.000,0.068) | 0.004(0.000,0.064) |
| 高风险组 | 427 | 0.000(0.000,0.228) | 0.024(0.000,0.480) | 0.139(0.003,0.793) | 0.053(0.000,0.239) | 0.004(0.000,0.116) | 0.000(0.000,0.083) | 0.010(0.000,0.053) |
| 检验统计量值 | 0.402 | -1.447 | -3.380 | 4.338 | -0.513 | -4.807 | -3.679 | |
| P值 | 0.69 | 0.15 | <0.01 | <0.01 | 0.61 | <0.01 | <0.01 | |
| 组别 | 滤泡辅助性T细胞 | 调节性T细胞 | γδT细胞 | 静息自然杀伤细胞 | 活化自然杀伤细胞 | 单核细胞 | M0型巨噬细胞 | |
| 低风险组 | 0.000(0.000,0.054) | 0.012(0.000,0.098) | 0.056(0.000,0.260) | 0.000(0.000,0.154) | 0.000(0.000,0.141) | 0.314(0.000,0.559) | 0.066(0.000,0.307) | |
| 高风险组 | 0.000(0.000,0.062) | 0.012(0.000,0.101) | 0.061(0.000,0.268) | 0.012(0.000,0.093) | 0.000(0.000,0.057) | 0.273(0.000,0.545) | 0.057(0.000,0.385) | |
| 检验统计量值 | 2.298 | 1.772 | -1.770 | -5.801 | 8.936 | 4.857 | 3.793 | |
| P值 | 0.02 | 0.08 | 0.08 | <0.01 | <0.01 | <0.01 | <0.01 | |
| 组别 | M1型巨噬细胞 | M2型巨噬细胞 | 静息树突状细胞 | 活化树突状细胞 | 静息肥大细胞 | 活化肥大细胞 | 嗜酸性粒细胞 | 中性粒细胞 |
| 低风险组 | 0.006(0.000,0.058) | 0.065(0.000,0.329) | 0.000(0.000,0.019) | 0.001(0.000,0.030) | 0.054(0.000,0.208) | 0.000(0.000,0.155) | 0.006(0.000,0.138) | 0.073(0.000,0.223) |
| 高风险组 | 0.006(0.000,0.149) | 0.069(0.000,0.373) | 0.000(0.000,0.072) | 0.003(0.000,0.037) | 0.042(0.000,0.244) | 0.002(0.000,0.143) | 0.008(0.000,0.142) | 0.073(0.000,0.318) |
| 检验统计量值 | 0.105 | -0.936 | -2.150 | -3.350 | 4.766 | -2.012 | -2.677 | 0.395 |
| P值 | 0.92 | 0.35 | 0.03 | <0.01 | <0.01 | 0.04 | <0.01 | 0.69 |
| 组别 | 例数 | HAVCR2 | CD274 | CD86 | LAG3 | LAIR1 | PVR |
|---|---|---|---|---|---|---|---|
| 低风险组 | 432 | 6.142(5.494,6.935) | 6.160(5.491,7.233) | 7.151(5.757,8.918) | 5.531(3.984,10.867) | 8.485(6.732,9.731) | 5.781(5.221,6.467) |
| 高风险组 | 427 | 6.048(5.334,8.257) | 6.282(5.518,8.503) | 7.212(5.142,9.660) | 5.506(4.207,8.743) | 8.470(6.030,9.795) | 5.642(5.230,6.316) |
| 检验统计量值 | 5.274 | -5.028 | -1.873 | -0.299 | 0.943 | 11.649 | |
| P值 | <0.01 | <0.01 | 0.06 | 0.77 | 0.35 | <0.01 | |
| 组别 | IDO1 | CD80 | CTLA4 | PDCD1 | TIGIT | CD200R1 | CEACAM1 |
| 低风险组 | 6.703(5.665,8.883) | 4.859(4.495,5.748) | 4.795(4.245,6.266) | 6.876(5.861,8.062) | 5.313(4.507,7.067) | 4.753(3.982,6.442) | 7.719(5.911,9.035) |
| 高风险组 | 6.676(5.393,11.180) | 4.798(4.449,6.503) | 4.670(4.230,5.660) | 6.563(5.580,7.892) | 5.326(4.182,6.711) | 4.863(4.070,7.110) | 7.733(5.552,9.270) |
| 检验统计量值 | 0.315 | 5.313 | 8.535 | 12.470 | -0.452 | -4.809 | 0.178 |
| P值 | 0.75 | <0.01 | <0.01 | <0.01 | 0.65 | <0.01 | 0.86 |
| 组别 | CD276 | CD200 | KIR3DL1 | BTLA | LGALS3 | VTCN1 | |
| 低风险组 | 7.418(6.750,8.190) | 7.036(5.482,10.333) | 4.434(3.888,6.273) | 6.796(5.419,10.172) | 12.208(9.402,13.108) | 4.489(3.850,5.611) | |
| 高风险组 | 7.306(6.564,8.361) | 7.649(5.969,12.027) | 4.412(3.919,5.514) | 7.419(5.179,12.785) | 11.882(9.575,13.005) | 4.399(3.736,5.874) | |
| 检验统计量值 | 5.732 | -8.090 | 1.604 | -6.797 | 9.584 | 4.467 | |
| P值 | <0.01 | <0.01 | 0.11 | <0.01 | <0.01 | <0.01 |
表9 不同风险组间免疫检查点基因的表达差异[M(P25,P75)]
Table 9 Expression differences of immune checkpoint genes among different risk groups
| 组别 | 例数 | HAVCR2 | CD274 | CD86 | LAG3 | LAIR1 | PVR |
|---|---|---|---|---|---|---|---|
| 低风险组 | 432 | 6.142(5.494,6.935) | 6.160(5.491,7.233) | 7.151(5.757,8.918) | 5.531(3.984,10.867) | 8.485(6.732,9.731) | 5.781(5.221,6.467) |
| 高风险组 | 427 | 6.048(5.334,8.257) | 6.282(5.518,8.503) | 7.212(5.142,9.660) | 5.506(4.207,8.743) | 8.470(6.030,9.795) | 5.642(5.230,6.316) |
| 检验统计量值 | 5.274 | -5.028 | -1.873 | -0.299 | 0.943 | 11.649 | |
| P值 | <0.01 | <0.01 | 0.06 | 0.77 | 0.35 | <0.01 | |
| 组别 | IDO1 | CD80 | CTLA4 | PDCD1 | TIGIT | CD200R1 | CEACAM1 |
| 低风险组 | 6.703(5.665,8.883) | 4.859(4.495,5.748) | 4.795(4.245,6.266) | 6.876(5.861,8.062) | 5.313(4.507,7.067) | 4.753(3.982,6.442) | 7.719(5.911,9.035) |
| 高风险组 | 6.676(5.393,11.180) | 4.798(4.449,6.503) | 4.670(4.230,5.660) | 6.563(5.580,7.892) | 5.326(4.182,6.711) | 4.863(4.070,7.110) | 7.733(5.552,9.270) |
| 检验统计量值 | 0.315 | 5.313 | 8.535 | 12.470 | -0.452 | -4.809 | 0.178 |
| P值 | 0.75 | <0.01 | <0.01 | <0.01 | 0.65 | <0.01 | 0.86 |
| 组别 | CD276 | CD200 | KIR3DL1 | BTLA | LGALS3 | VTCN1 | |
| 低风险组 | 7.418(6.750,8.190) | 7.036(5.482,10.333) | 4.434(3.888,6.273) | 6.796(5.419,10.172) | 12.208(9.402,13.108) | 4.489(3.850,5.611) | |
| 高风险组 | 7.306(6.564,8.361) | 7.649(5.969,12.027) | 4.412(3.919,5.514) | 7.419(5.179,12.785) | 11.882(9.575,13.005) | 4.399(3.736,5.874) | |
| 检验统计量值 | 5.732 | -8.090 | 1.604 | -6.797 | 9.584 | 4.467 | |
| P值 | <0.01 | <0.01 | 0.11 | <0.01 | <0.01 | <0.01 |
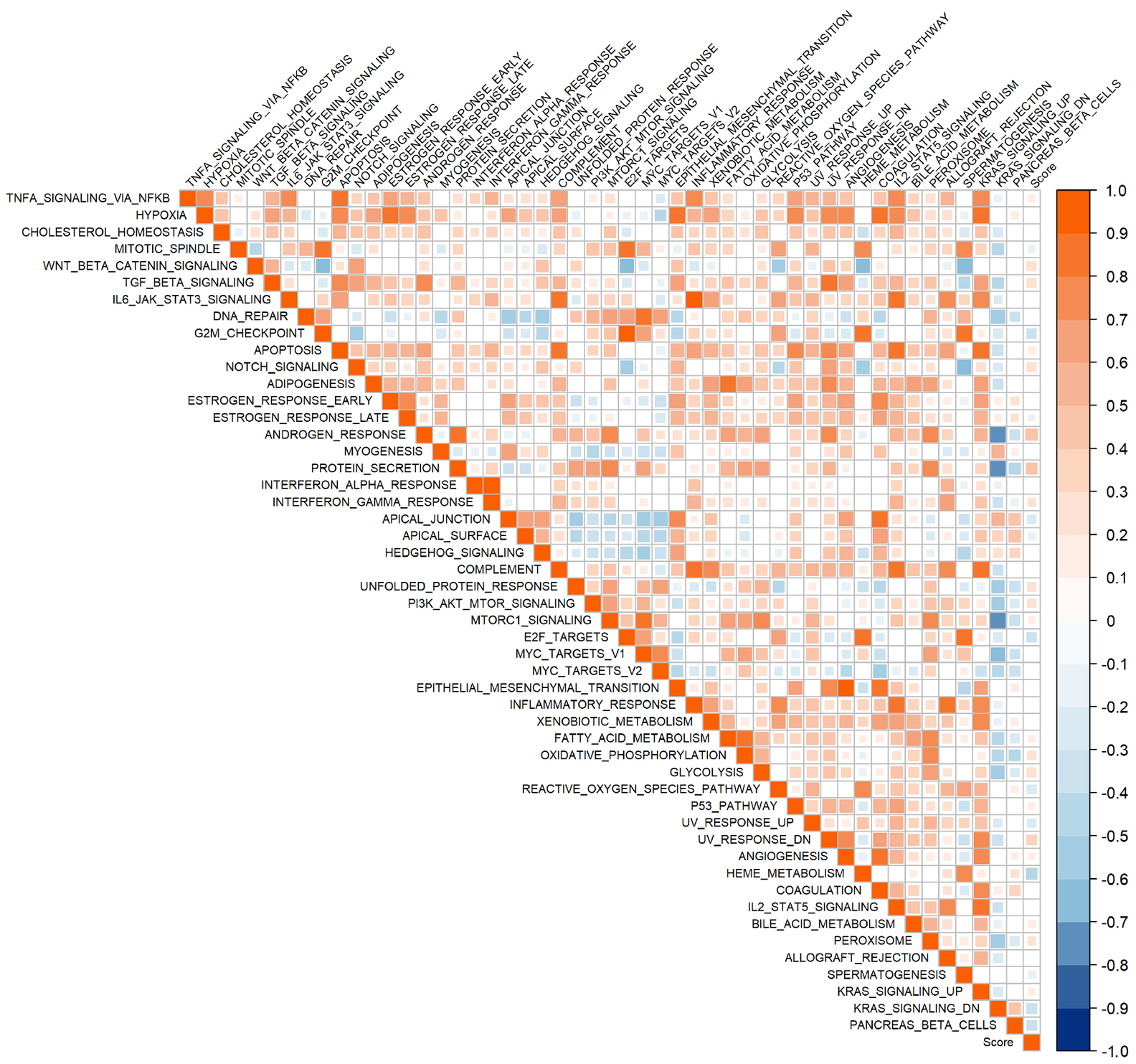
图14 乳酸代谢预后模型风险得分与50 hallmark通路的相关性分析
Figure 14 Correlation between the risk score of the lactate metabolism prognostic model and the 50 hallmark pathway
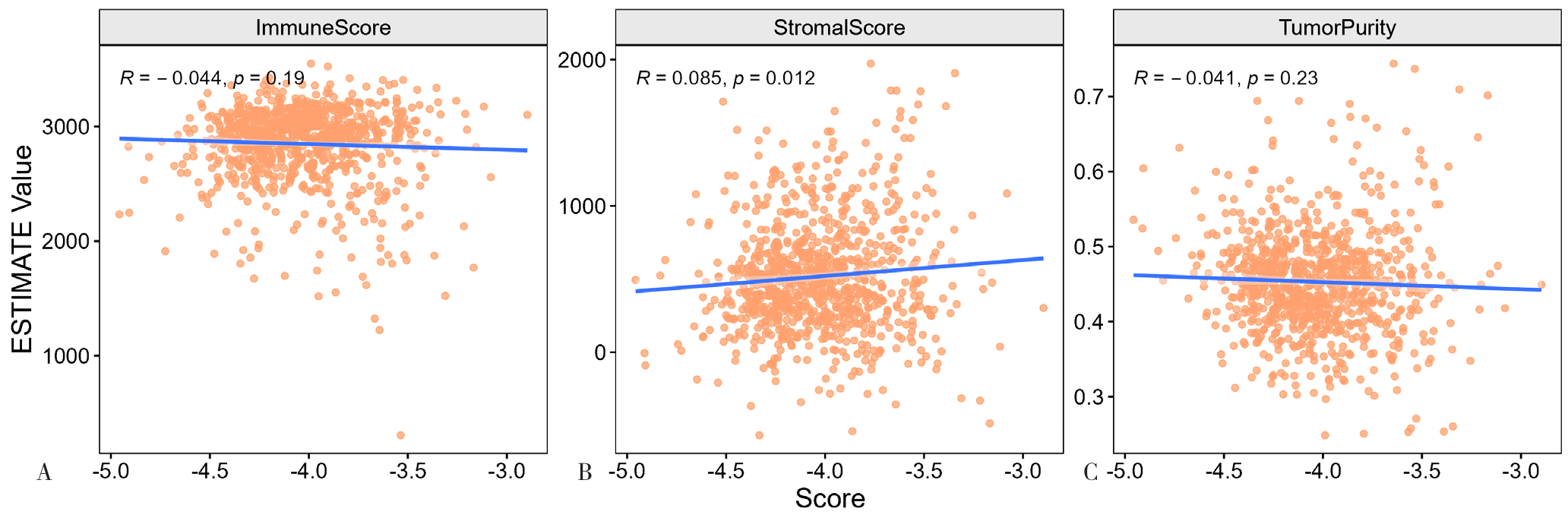
图15 乳酸代谢预后模型风险得分与免疫评分、基质评分和肿瘤纯度的相关性分析注:A为乳酸代谢预后模型风险得分与免疫评分的相关性,B为乳酸代谢预后模型风险得分与基质评分的相关性,C为乳酸代谢预后模型风险得分与肿瘤纯度的相关性。
Figure 15 Correlation analysis of the risk score of the lactate metabolism prognosis model with the immune score, matrix score and tumor purity
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [1] | 郑博月, 付积艺, 吴佳霏, 王珺, 李慧. 卡非佐米治疗多发性骨髓瘤的疗效及安全性研究[J]. 中国全科医学, 2025, 28(30): 3806-3814. |
| [2] | 阮万百, 李俊峰, 尹艳梅, 彭磊, 朱克祥. 胰腺癌靶向治疗及免疫治疗的研究新进展[J]. 中国全科医学, 2025, 28(23): 2950-2960. |
| [3] | 陈飞, 王金英, 于海搏, 李新, 张佳佳, 申曼, 詹晓凯, 汤然, 范斯斌, 赵凤仪, 张天宇, 黄仲夏. 中性粒细胞明胶酶相关运载蛋白、T细胞免疫球蛋白粘蛋白受体1、血管细胞黏附分子-1和激活素A升高在新诊断多发性骨髓瘤患者中的意义研究[J]. 中国全科医学, 2025, 28(22): 2740-2749. |
| [4] | 赵凤仪, 李新, 詹晓凯, 张佳佳, 申曼, 汤然, 范斯斌, 黄仲夏. 初诊超高龄多发性骨髓瘤患者硼替佐米基础方案治疗的生存预后分析[J]. 中国全科医学, 2024, 27(08): 971-977. |
| [5] | 于海搏, 张天宇, 李新, 张佳佳, 申曼, 詹晓凯, 汤然, 范斯斌, 赵凤仪, 黄仲夏. 双靶点嵌合抗原受体-T细胞治疗复发难治多发性骨髓瘤患者疗效和安全性的Meta分析[J]. 中国全科医学, 2024, 27(08): 985-994. |
| [6] | 彭逸伦, 李杨, 王晓桃. 多发性骨髓瘤细胞通过PI3K/AKT信号通路促进M2巨噬细胞极化的机制研究[J]. 中国全科医学, 2024, 27(08): 978-984. |
| [7] | 裴蓓, 成琳, 许凌云. 不同新辅助化疗方案对人表皮生长因子受体2阳性乳腺癌患者免疫指标和肿瘤微环境的影响研究[J]. 中国全科医学, 2023, 26(27): 3435-3440. |
| [8] | 王珺, 吴佳霏, 王依景, 郑博月, 王宇, 江川艳, 李慧. 以达雷妥尤单抗为基础的化疗方案对多发性骨髓瘤疗效和预后影响的真实世界研究[J]. 中国全科医学, 2023, 26(18): 2256-2262. |
| [9] | 劳深, 何建行, 梁文华. 靶向周细胞肿瘤血管正常化:重塑肿瘤微环境的新策略[J]. 中国全科医学, 2022, 25(32): 3971-3977. |
| [10] | 李科, 李征, 耿惠, 马婕. CD38单克隆抗体治疗多发性骨髓瘤疗效和安全性的Meta分析[J]. 中国全科医学, 2022, 25(21): 2661-2669. |
| [11] | 杨婧诗,邹立群. 淋巴瘤相关静脉血栓的影响因素及风险评估模型的研究进展[J]. 中国全科医学, 2021, 24(23): 2992-2997. |
| [12] | 裴晓姣,潘振宇,炼宇飞,申曼,蒋涛,黄仲夏. 椎体骨髓脂肪含量比在新诊断多发性骨髓瘤中的诊断价值[J]. 中国全科医学, 2021, 24(20): 2601-2606. |
| [13] | 项秋晴,陆敏秋,褚彬,王宇彤,石磊,高珊,房立娟,刘晰,丁月华,鲍立. 达雷妥尤单抗治疗复发/难治性多发性骨髓瘤疗效观察研究[J]. 中国全科医学, 2021, 24(2): 237-242. |
| [14] | 王腾,王晓晨,吕纯懿,王金鑫,徐瑞荣. 嵌合抗原受体T淋巴细胞治疗多发性骨髓瘤疗效及安全性的Meta分析[J]. 中国全科医学, 2021, 24(2): 219-224. |
| [15] | 申曼,黄仲夏,李新,张佳佳,裴晓姣,潘振宇,陈文明. 硼替佐米治疗新诊断多发性骨髓瘤期间心脏不良事件的影响因素及其对生存时间影响的真实世界研究[J]. 中国全科医学, 2021, 24(2): 210-218. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||





