Page 47 - 2023-08-中国全科医学
P. 47
·944· http: //www.chinagp.net E-mail: zgqkyx@chinagp.net.cn March 2023, Vol.26 No.8
-3
损伤刺激反应、单纯疱疹病毒 1 型感染、RNA 代谢、 P=4.2×10 ) 模 块 呈 正 相 关, 与 deeppink(r=0.50,
-3
调节细胞对压力的反应、适应性免疫系统等,TRRUST P=0.01)、lavender(r=0.60,P=2.6×10 )、
-3
数据库预测 MHC Ⅱ反式激活蛋白(CIITA)转录因子可 lavenderblush3(r=0.56,P=5.9×10 )呈负相关,以
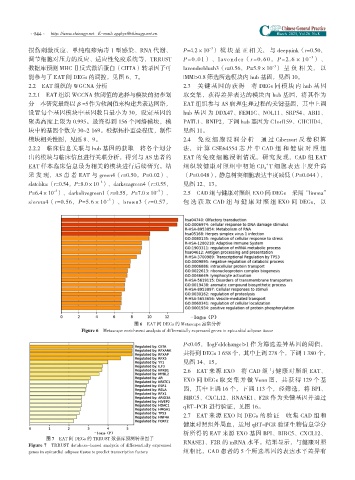
能参与了 EAT 间 DEGs 的调控,见图 6、7。 |MM|>0.8 筛选所选模块内 hub 基因,见图 10。
2.2 EAT 组织的 WGCNA 分析 2.3 关键基因的获得 将 DEGs 同模块内 hub 基因
2.2.1 EAT 组织 WGCNA 软阈值的选择与模块的初步划 取交集,获得差异表达的模块内 hub 基因,将其作为
分 本研究最终以β=5作为软阈值来构建共表达网络, EAT 组织参与 AS 病理生理过程的关键基因,其中上调
设置每个基因模块中基因数目最小为 30,设定基因的 hub 基 因 为 DDX47、FEM1C、NOL11、SRP54、ABI1、
聚类高度上限为 0.995。最终得到 156 个网络模块,模 PATL1、BNIP2,下调 hub 基因为 C1orf159、CHCHD4,
块中的基因个数为 30~2 169。根据拓扑重叠程度,制作 见图 11。
模块相关性图,见图 8、9。 2.4 免疫细胞浸润分析 通过 Cibersort 反卷积算
2.2.2 临床信息关联与 hub 基因的获取 将各个划分 法, 计 算 GSE64554 芯 片 中 CAD 组 和 健 康 对 照 组
出的模块与临床信息进行关联分析,得到与 AS 患者的 EAT 的免疫细胞浸润情况。研究发现,CAD 组 EAT
+
EAT 样本临床信息最为相关的模块进行后续研究。结 组织较健康对照组中初始 CD 4 T 细胞表达丰度升高
果 发 现,AS 患 者 EAT 与 green4(r=0.50,P=0.02)、 (P=0.048),静息树突细胞表达丰度减低(P=0.044),
-3
slateblue(r=0.54,P=8.0×10 )、darkseagreen4(r=0.55, 见图 12、13。
-3
-3
P=6.4×10 )、darkolivegreen1(r=0.55,P=7.0×10 )、 2.5 CAD 组与健康对照组 EXO 间 DEGs 采用“limma”
-3
sienna4(r=0.56,P=5.6×10 )、brown3(r=0.57, 包选获取 CAD 组与健康对照组 EXO 间 DEGs,以
图 6 EAT 间 DEGs 的 Metascape 富集分析
Figure 6 Metascape enrichment analysis of differentially expressed genes in epicardial adipose tissue
P<0.05,|logFoldchange|>1 作为筛选差异基因的阈值,
共得到 DEGs 1 658 个,其中上调 278 个,下调 1 380 个,
见图 14、15。
2.6 EAT 来源 EXO 将 CAD 组与健康对照组 EAT、
EXO 间 DEGs 取 交 集 并 做 Venn 图, 共 获 得 129 个 基
因,其中上调 16 个,下调 113 个,经筛选,将 BPI、
BIRC5、CXCL12、RNASE1、F2R 作为关键基因并通过
qRT-PCR 进行验证,见图 16。
2.7 EAT 来源 EXO 间 DEGs 的验证 收集 CAD 组和
健康对照组外周血,运用 qRT-PCR 验证生物信息学分
析所得的 EAT 来源 EXO 基因 BPI、BIRC5、CXCL12、
图 7 EAT 间 DEGs 的 TRRUST 数据库预测转录因子 RNASE1、F2R 的 mRNA 水平。结果显示,与健康对照
Figure 7 TRRUST database-based analysis of differentially expressed
genes in epicardial adipose tissue to predict transcription factors 组相比,CAD 患者的 5 个所选基因的表达水平差异有