Page 92 - 中国全科医学2022-14
P. 92
http://www.chinagp.net E-mail:zgqkyx@chinagp.net.cn ·1753·
矩估计法(Adaptive Moment Estimation,Adam) [23] 进 别信息,和样本级分类损失的联合优化在病毒形态诊断
行优化,其中学习率为 1e-5 和权重衰减率为 5e-4。经 任务中可以获得更好的结果。见表 3。
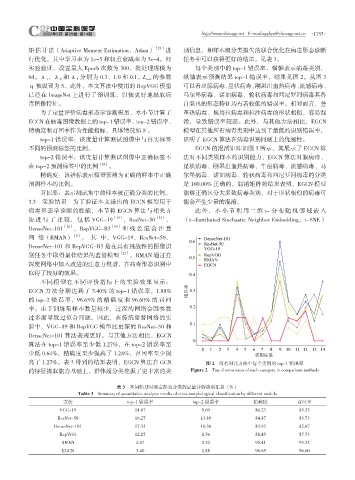
实验验证,设置最大 Epoch 次数为 300,批处理规模为 每个类别中的 top-1 错误率,横轴表示病毒类别,
64。λ 1 ,λ 2 和 λ 3 分别为 0.3、1.0 和 0.1。L com 的参数 纵轴表示预测结果 top-1 错误率,结果见图 2。从图 3
η 被设置为 5。此外,本文算法中使用的 RepVGG 模型 可以看出腺病毒、星状病毒、刚果出血热病毒、流感病毒、
已经在 ImageNet 上进行了预训练,以便更好地提取病 马尔堡病毒、诺如病毒、轮状病毒和西尼罗河病毒其各
毒图像特征。 自突出的形态特征均有着较低的错误率。相对而言,登
为了定量评价病毒形态学诊断模型,本小节计算了 革热病毒、埃博拉病毒和拉沙病毒的形状相似,容易混
EGCN 在病毒图像数据上的 top-1 错误率、top-2 错误率、 淆,导致错误率较高。此外,与其他方法相比,EGCN
精确度和召回率作为性能指标,具体情况如下。 模型在其他所有病毒类别中达到了最低的识别错误率,
top-1 错误率:该度量计算测试图像中与真实标签 证明了 EGCN 算法在病毒识别问题上的优越性。
不同的预测标签的比例。 EGCN 的混淆矩阵如图 3 所示,其展示了 EGCN 算
top-2 错误率:该度量计算测试图像中正确标签不 法对不同类别样本的识别能力,EGCN 算法对腺病毒、
在 top-2 预测标签中的比例 [24] 。 星状病毒、刚果出血热病毒、牛痘病毒、流感病毒、马
精确度:该指标表示模型预测为正确的样本中正确 尔堡病毒、诺如病毒、轮状病毒和西尼罗河病毒的分类
预测样本的比例。 是 100.00% 正确的。混淆矩阵的结果表明,EGCN 模型
召回率:表示测试集中的样本被正确分类的比例。 能够正确区分大多数病毒类别,对于形状相似的病毒可
3.3 实验结果 为了验证本文提出的 EGCN 模型用于 能会产生少量的混淆。
病毒形态学诊断的性能,本节将 EGCN 算法与相关方 此外,本小节利用二维 t- 分布随机邻域嵌入
法进行了比较,包括 VGG-19 [19] ,ResNet-50 [25] , (t-distributed Stochastic Neighbor Embedding,t-SNE)
DenseNet-101 [26] ,RepVGG-B3 [20] 和 残差 混合 注意
网 络(RMAN) [6] 。 其 中,VGG-19,ResNet-50,
DenseNet-101 和 RepVGG-B3 是在具有挑战性的图像识
别任务中取得最佳结果的监督模型 [27] 。RMAN 通过在
深度网络中加入改进的注意力模型,在病毒形态识别中
取得了较好的效果。
不同模型在不同评价指标上的实验效果显示:
EGCN 方法分别达到了 3.40% 的 top-1 错误率,1.88%
的 top-2 错 误 率,96.65% 的 精 确 度 和 96.60% 的 召 回
率。由于训练集样本数量较少,过深的网络会因参数
过多而导致过拟合问题。因此,在传统监督网络的实
验中,VGG-19 和 RepVGG 模型比更深的 ResNet-50 和
DenseNet-101 算法表现更好。与其他方法相比,EGCN
算法在 top-1 错误率至少低 1.27%,在 top-2 错误率至
少低 0.64%,精确度至少提高了 1.24%,召回率至少提
高了 1.27%。表 3 得到的结果表明,EGCN 算法在 GCN 图 2 所有对比方法中每个类别的 top-1 错误率
的特征提取能力基础上,群体超分类挖掘了更丰富的类 Figure 2 Top-1 error rates of each category in comparison methods
表 3 不同模型对病毒形态分类的定量分析结果汇总(%)
Table 3 Summary of quantitative analysis results of virus morphological classification by different models
方法 top-1 错误率 top-2 错误率 精确度 召回率
VGG-19 14.67 9.69 86.23 85.33
ResNet-50 16.27 13.89 84.87 83.73
DenseNet-101 17.33 10.36 83.93 82.67
RepVGG 12.27 8.56 88.45 87.73
RMAN 4.67 2.52 95.41 95.33
EGCN 3.40 1.88 96.65 96.60